Questo è il secondo articolo parte della serie dedicata alla network science. Puoi leggere il primo articolo, intitolato Introduzione alla Network Analysis in Python: Pulizia, Analisi e Visualizzazione di reti di grafi per iniziare il tuo percorso nello studio dei grafi di rete.
Tabella dei Contenuti
Le reti sono ovunque: reti sociali, reti di trasporto, reti biologiche, reti di transazioni. E, nella realtà, queste reti non sono statiche. Gli utenti si iscrivono o abbandonano piattaforme, i contatti sociali cambiano, i dispositivi si connettono e si disconnettono. Per questo motivo, parlare oggi di “reti” senza considerare la loro evoluzione nel tempo significa trascurare una dimensione cruciale. Da qui nasce l’interesse per le reti dinamiche: strutture che cambiano nel tempo, e che richiedono metodi specifici per essere analizzate.
Uno degli obiettivi principali nello studio delle reti dinamiche è la link prediction: la previsione dei collegamenti futuri tra i nodi.
Questa domanda è centrale in diversi contesti:
- nelle piattaforme sociali, per suggerire nuove amicizie;
- nella biologia computazionale, per ipotizzare nuove interazioni tra proteine;
- nei sistemi di raccomandazione, per prevedere quali utenti interagiranno con quali prodotti.
Il problema della link prediction non è nuovo. Ma diventa particolarmente sfidante nel contesto dinamico, dove la struttura della rete cambia nel tempo e le informazioni disponibili possono essere parziali o rumorose. Per affrontarlo servono approcci teorici solidi e implementazioni efficienti.
1. chiarire cosa significa lavorare con grafi dinamici;
2. distinguere tra diverse strategie di modellazione temporale (snapshot vs eventi con timestamp);
3. mostrare come funziona la link prediction, sia con approcci euristici (basati sulla similarità tra nodi) che con modelli supervisionati;
4. applicare queste tecniche a un caso reale, usando un dataset pubblico e librerie Python open-source.
Con l’aumento delle reti temporali (dati da sensori, log di attività, interazioni digitali), saper modellare e anticipare l’evoluzione delle connessioni è sempre più importante. La link prediction non è solo un esercizio teorico: è uno strumento utile per potenziare modelli predittivi, raccomandazioni intelligenti e politiche di intervento mirato nei sistemi complessi.
Reti dinamiche: definizione e modelli
Le reti dinamiche rappresentano un’estensione naturale dei grafi statici per modellare fenomeni che evolvono nel tempo. Per catturare questa variabilità temporale, la teoria delle reti ha introdotto i concetti di time-varying graphs o dynamic networks.
Una rete dinamica è un grafo i cui nodi e archi possono comparire, sparire o modificarsi nel tempo. Formalmente, è una sequenza ordinata di grafi \( G_t = (V_t, E_t) \) indicizzati da un tempo discreto \( \text{t} \). Ogni snapshot \( G_t \) rappresenta lo stato del sistema in un dato intervallo temporale. In alternativa, una rete dinamica può essere modellata come un insieme di eventi con un timestamp associato, dove ciascun evento rappresenta un'interazione istantanea (ad esempio, un contatto fisico tra due studenti).
Questa struttura rende le reti dinamiche più complesse ma anche più aderenti ai dati reali rispetto ai grafi statici.
Due approcci di modellazione: snapshot vs time-edges
Esistono due principali strategie per rappresentare reti dinamiche:
- Modello a snapshot (o grafi a intervalli):
- Si discretizza il tempo in intervalli (es. ogni ora, giorno, minuto).
- Si costruisce un grafo statico per ciascun intervallo.
- Ogni snapshot contiene tutti gli archi attivi in quell’intervallo.
- È utile per analisi basate su finestre temporali (sliding windows, aggregazioni).
- Limite: la scelta della granularità influenza fortemente i risultati.
- Modello stream-based (o time-edges):
- Ogni arco è un evento associato a un timestamp specifico.
- Non c’è aggregazione: si mantiene il dettaglio completo della dinamica.
- Consente analisi più precise, ma richiede maggiore potenza computazionale.
- È adatto quando gli eventi sono frequenti e granulari (es. contatti ogni 20s nel dataset SocioPatterns).
Entrambi i modelli hanno vantaggi e limiti. La scelta dipende dallo scopo dell’analisi: aggregazioni e pattern stabili si adattano meglio agli snapshot; processi di diffusione o previsioni fine-grained richiedono la modellazione stream-based.
Riflessioni pratiche
Le reti dinamiche introducono nuove complessità rispetto ai grafi statici:
- Persistenza dei nodi: i nodi possono essere presenti in alcuni snapshot e assenti in altri.
- Evoluzione topologica: metriche classiche (grado, centralità, clustering coefficient) vanno ricalcolate a ogni intervallo.
- Analisi temporale: è possibile tracciare come evolve la posizione di un nodo nella rete (es. nodo periferico → centrale).
Queste proprietà permettono di affrontare problemi avanzati come la link prediction temporale, ovvero la previsione di connessioni che potrebbero comparire nel futuro. In questo contesto, la rete dinamica non è solo un supporto descrittivo, ma diventa la base per modelli predittivi.
Il Dataset: High School Contact Network
Per analizzare l’evoluzione delle reti nel tempo, useremo il dataset High School Contact Network, reso disponibile dal progetto SocioPatterns. Questo dataset è ideale per lo studio delle reti dinamiche perché fornisce una rappresentazione ad alta risoluzione temporale dei contatti fisici tra studenti in un ambiente scolastico reale.
Struttura del dataset
Ogni riga rappresenta un’interazione tra due studenti rilevata in un intervallo di 20 secondi, con le seguenti colonne:
| Colonna | Significato |
|---|---|
t | Timestamp in secondi UNIX |
i | ID anonimo dello studente 1 |
j | ID anonimo dello studente 2 |
Ci | Classe dello studente 1 |
Cj | Classe dello studente 2 |
Il contatto è attivo tra t - 20 e t secondi. Se più contatti sono attivi nello stesso istante, ci sono più righe con lo stesso t.
Accesso al dataset in Python
Il dataset non è disponibile via librerie come networkx o dynetx. Va quindi scaricato e caricato manualmente:
import pandas as pd
import urllib.request
import gzip
from io import BytesIO
# URL del file
url = "http://www.sociopatterns.org/wp-content/uploads/2015/07/High-School_data_2013.csv.gz"
# Scarica e leggi usando separatore whitespace
with urllib.request.urlopen(url) as response:
with gzip.GzipFile(fileobj=BytesIO(response.read())) as f:
df = pd.read_csv(f, delim_whitespace=True, header=None,
names=['timestamp', 'i', 'j', 'Ci', 'Cj'])
# Controllo tipi
print(df.dtypes)
print(df.head())
>>>
timestamp int64
i int64
j int64
Ci object
Cj object
dtype: object
timestamp i j Ci Cj
0 1385982020 454 640 MP MP
1 1385982020 1 939 2BIO3 2BIO3
2 1385982020 185 258 PC* PC*
3 1385982020 55 170 2BIO3 2BIO3
4 1385982020 9 453 PC PC
Statistiche descrittive
print("Numero di contatti unici:", df.shape[0])
print("Numero di studenti:", len(set(df['i']).union(set(df['j']))))
print("Intervallo temporale (sec):", df['timestamp'].min(), "→", df['timestamp'].max())
>>>
Numero di contatti unici: 188508
Numero di studenti: 327
Intervallo temporale (sec): 1385982020 → 1386345580Costruzione della rete dinamica
Una rete dinamica può essere rappresentata in due modi:
- Snapshot: una serie di grafi statici a intervalli regolari (es. ogni 15 minuti).
- Stream: una sequenza di archi con timestamp.
Qui adotteremo l’approccio snapshot-based.
Creazione snapshot
Ogni snapshot corrisponde a una finestra temporale aggregata (es. 15 minuti = 900 secondi). Costruiamo una lista di grafi, uno per ciascun intervallo.
import networkx as nx
from collections import defaultdict
# Parametri
delta = 900 # Durata snapshot in secondi (15 minuti)
start = df['timestamp'].min()
end = df['timestamp'].max()
# Costruzione snapshot
snapshots = []
for t in range(start, end, delta):
df_window = df[(df['timestamp'] >= t) & (df['timestamp'] < t + delta)]
G = nx.Graph()
G.add_edges_from(zip(df_window['i'], df_window['j']))
snapshots.append(G)
print(f"Numero di snapshot creati: {len(snapshots)}")
>>>
Numero di snapshot creati: 404Grafo aggregato
Per una prima visualizzazione esplorativa, costruiamo il grafo aggregato, ossia la rete totale risultante dall’unione di tutti i contatti.
G_total = nx.Graph()
G_total.add_edges_from(zip(df['i'], df['j']))
print("Numero di nodi aggregati:", G_total.number_of_nodes())
print("Numero di archi aggregati:", G_total.number_of_edges())
>>>
Numero di nodi aggregati: 327
Numero di archi aggregati: 5818Visualizzazione della rete aggregata
import matplotlib.pyplot as plt
plt.figure(figsize=(12, 8))
pos = nx.spring_layout(G_total, seed=42)
nx.draw_networkx_nodes(G_total, pos, node_size=30, alpha=0.7)
nx.draw_networkx_edges(G_total, pos, alpha=0.2)
plt.title("Grafo Aggregato dei Contatti (High School)")
plt.axis('off')
plt.show()
Analisi preliminare dei nodi
Calcoliamo alcune metriche base di centralità sul grafo aggregato, utili come feature per modelli supervisionati di link prediction.
degree_dict = dict(G_total.degree())
closeness_dict = nx.closeness_centrality(G_total)
betweenness_dict = nx.betweenness_centrality(G_total, normalized=True)
# Trasformazione in DataFrame
df_nodes = pd.DataFrame({
'node': list(G_total.nodes()),
'degree': [degree_dict[n] for n in G_total.nodes()],
'closeness': [closeness_dict[n] for n in G_total.nodes()],
'betweenness': [betweenness_dict[n] for n in G_total.nodes()],
})
print(df_nodes.head())
>>>
node degree closeness betweenness
0 454 26 0.442935 0.000614
1 640 29 0.445355 0.001557
2 1 23 0.425587 0.000864
3 939 36 0.443537 0.000971
4 185 43 0.503864 0.004479
Network Science with Python
David Knickerbocker
Guida hands‑on di network science in Python, con un solido mix di teoria, esempi pratici e integrazione NLP + ML, pensata per chi vuole applicare l'analisi delle reti a problemi concreti.
Estrazione di feature topologiche
Le tecniche di link prediction supervisionata richiedono feature sui nodi e sulle coppie. Iniziamo calcolando alcune metriche base sul grafo aggregato:
# Metriche di centralità
degree_dict = dict(G_total.degree())
closeness_dict = nx.closeness_centrality(G_total)
betweenness_dict = nx.betweenness_centrality(G_total, normalized=True)
# Costruzione dataframe nodi
df_nodes = pd.DataFrame({
'node': list(G_total.nodes()),
'degree': [degree_dict[n] for n in G_total.nodes()],
'closeness': [closeness_dict[n] for n in G_total.nodes()],
'betweenness': [betweenness_dict[n] for n in G_total.nodes()],
})
print(df_nodes.head())
>>>
node degree closeness betweenness
0 454 26 0.442935 0.000614
1 640 29 0.445355 0.001557
2 1 23 0.425587 0.000864
3 939 36 0.443537 0.000971
4 185 43 0.503864 0.004479Matrici di adiacenza per snapshot
Ogni snapshot può essere convertito in matrice di adiacenza per l’analisi o la costruzione di feature per modelli supervisionati:
import numpy as np
# Lista completa dei nodi (fissa per tutti gli snapshot)
all_nodes = sorted(G_total.nodes())
# Lista di matrici di adiacenza coerenti
adj_matrices = []
for G in snapshots:
G_full = nx.Graph()
G_full.add_nodes_from(all_nodes) # garantisce presenza di tutti i nodi
G_full.add_edges_from(G.edges()) # aggiunge solo gli archi dello snapshot corrente
A = nx.to_numpy_array(G_full, nodelist=all_nodes)
adj_matrices.append(A)
Link prediction: concetti chiave
La link prediction è una tecnica usata per prevedere quali nuove connessioni tra nodi compariranno in futuro. Si applica a contesti diversi: reti sociali (amicizie che si formeranno), reti biologiche (interazioni proteiche sconosciute), reti finanziarie (transazioni potenziali), ecc.
Nel contesto di reti dinamiche, l’obiettivo è predire se due nodi saranno collegati nello snapshot futuro, usando informazioni dagli snapshot precedenti.
Approcci principali
Esistono due famiglie principali di approcci:
1. Approcci non supervisionati
- Si basano su misure di similarità tra nodi già presenti nella rete.
- Non richiedono modelli di apprendimento.
- Esempi:
- Common Neighbors: numero di vicini in comune
- Jaccard Coefficient: rapporto tra vicini in comune e unione dei vicini
- Adamic-Adar: penalizza i vicini comuni molto connessi
2. Approcci supervisionati
- Si costruisce un dataset di training con coppie di nodi e feature strutturali (es. centralità, similarità).
- Si etichettano le coppie come 1 (comparsa del link nello snapshot successivo) o 0 (nessun link).
- Si applicano modelli ML (es. Random Forest, Logistic Regression).
Metriche di valutazione
La predizione di link è un problema sbilanciato (pochi link rispetto alle non-connessioni). Le metriche più usate:
| Metrica | Significato |
|---|---|
| AUC | Area under ROC curve: capacità di distinguere i positivi |
| Precision@k | Percentuale di link correttamente predetti nei top-k |
| Recall@k | Percentuale di link reali trovati nei top-k predetti |
Quando usare l'uno o l'altro:
| Approccio | Vantaggi | Limiti |
|---|---|---|
| Non supervisionato | Semplice, rapido | Poco flessibile, no apprendimento |
| Supervisionato | Alta precisione, estensibile | Serve dataset bilanciato e feature |
Tecniche di link prediction: implementazione
Partiamo con una prima implementazione su snapshot consecutivi, usando approcci non supervisionati. In seguito vedremo il modello supervisionato.
Metodo 1: Similarità locale (Common Neighbors, Jaccard, Adamic-Adar)
Prendiamo due snapshot consecutivi e calcoliamo le similarità tra tutte le coppie di nodi non collegate nel primo snapshot, ma che potrebbero collegarsi nel secondo.
import networkx as nx
from networkx.algorithms.link_prediction import (
jaccard_coefficient,
adamic_adar_index,
preferential_attachment
)
# Scegli snapshot consecutivi
G_train = snapshots[100]
G_test = snapshots[101]
# Nodi presenti nello snapshot
nodes = list(G_train.nodes())
# Coppie potenziali (non ancora collegate)
potential_edges = [
(u, v) for i, u in enumerate(nodes) for v in nodes[i + 1:]
if not G_train.has_edge(u, v)
]
# Common Neighbors (manuale)
def compute_common_neighbors(G, pairs):
return [(u, v, len(list(nx.common_neighbors(G, u, v)))) for u, v in pairs]
cn_scores = compute_common_neighbors(G_train, potential_edges)
# Jaccard
jaccard_scores = list(jaccard_coefficient(G_train, potential_edges))
# Adamic-Adar
aa_scores = list(adamic_adar_index(G_train, potential_edges))
# Preferential Attachment (opzionale)
pa_scores = list(preferential_attachment(G_train, potential_edges))
def label_links(pairs, G_future):
return [1 if G_future.has_edge(u, v) else 0 for u, v in pairs]
labels_cn = label_links([(u, v) for u, v, _ in cn_scores], G_test)
labels_jaccard = label_links([(u, v) for u, v, _ in jaccard_scores], G_test)
labels_aa = label_links([(u, v) for u, v, _ in aa_scores], G_test)
Valutazione AUC per Jaccard:
from sklearn.metrics import roc_auc_score
jaccard_values = [score for _, _, score in jaccard_scores]
auc_jaccard = roc_auc_score(labels_jaccard, jaccard_values)
print(f"AUC (Jaccard): {auc_jaccard:.4f}")
>>>
AUC (Jaccard): 0.6466Modello supervisionato per link prediction
Il metodo supervisionato richiede:
- Coppie di nodi candidate (potenziali link futuri)
- Etichette binarie: 1 se il link compare nel prossimo snapshot, 0 altrimenti
- Feature strutturali per ogni coppia: common neighbors, jaccard, adamic-adar, etc.
Step 1: Definizione snapshot
Scegliamo due snapshot consecutivi per costruire il training set:
G_train = snapshots[100]
G_test = snapshots[101]
nodes = list(G_train.nodes())
Step 2: Costruzione delle coppie e delle label
# Tutte le coppie non connesse nello snapshot corrente
candidate_pairs = [
(u, v) for i, u in enumerate(nodes) for v in nodes[i + 1:]
if not G_train.has_edge(u, v)
]
# Label binaria: 1 se (u,v) è presente in G_test, altrimenti 0
def label_links(pairs, G_future):
return [1 if G_future.has_edge(u, v) else 0 for u, v in pairs]
labels = label_links(candidate_pairs, G_test)Step 3: Estrazione delle feature
# Funzione per estrarre tutte le feature strutturali
def extract_features(G, pairs):
cn = { (u,v): len(list(nx.common_neighbors(G, u, v))) for u,v in pairs }
jc = { (u,v): p for u,v,p in jaccard_coefficient(G, pairs) }
aa = { (u,v): p for u,v,p in adamic_adar_index(G, pairs) }
pa = { (u,v): p for u,v,p in preferential_attachment(G, pairs) }
data = []
for u, v in pairs:
data.append({
'u': u,
'v': v,
'cn': cn.get((u,v), 0),
'jc': jc.get((u,v), 0),
'aa': aa.get((u,v), 0),
'pa': pa.get((u,v), 0),
})
return pd.DataFrame(data)
df_features = extract_features(G_train, candidate_pairs)
df_features['label'] = labels
print(df_features.head())
>>>
u v cn jc aa pa label
0 61 1345 0 0.0 0.0 24 0
1 61 1401 0 0.0 0.0 39 0
2 61 1828 0 0.0 0.0 12 0
3 61 196 0 0.0 0.0 12 0
4 61 779 0 0.0 0.0 6 0Step 4: Addestramento modello
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import roc_auc_score, precision_score
X = df_features[['cn', 'jc', 'aa', 'pa']]
y = df_features['label']
# Train/test split
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, test_size=0.3, random_state=42)
# Random Forest
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
# Valutazione
y_pred = clf.predict(X_test)
y_prob = clf.predict_proba(X_test)[:, 1]
print("AUC:", roc_auc_score(y_test, y_prob))
print("Precision:", precision_score(y_test, y_pred))
>>>
AUC: 0.5936154107175485
Precision: 0.6666666666666666Step 5: Analisi delle feature importanti
import matplotlib.pyplot as plt
importances = clf.feature_importances_
plt.bar(X.columns, importances)
plt.title("Importanza delle feature per il modello")
plt.ylabel("Peso")
plt.show()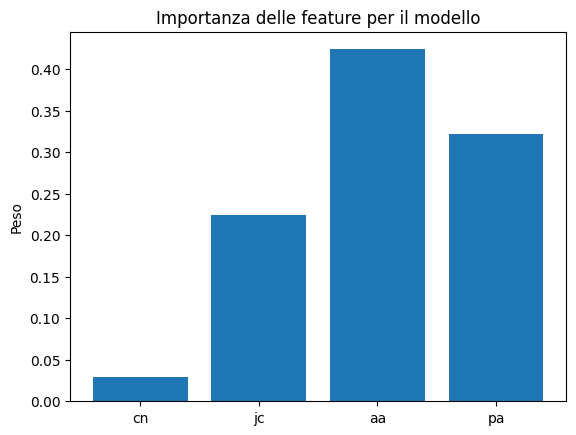
Visualizzazione dinamica
Creiamo un’animazione della rete nei primi 20 snapshot per mostrare l’evoluzione temporale.
import matplotlib.pyplot as plt
import matplotlib.animation as animation
import networkx as nx
# Fissa i nodi su un layout globale (grafico aggregato)
pos = nx.spring_layout(G_total, seed=42)
# Prepara la figura
fig, ax = plt.subplots(figsize=(10, 8))
def draw_snapshot(i):
ax.clear()
G = snapshots[i]
nx.draw_networkx_nodes(G, pos, ax=ax, node_size=30, alpha=0.7)
nx.draw_networkx_edges(G, pos, ax=ax, alpha=0.2)
ax.set_title(f"Snapshot {i}")
ax.axis('off')
# Genera animazione
ani = animation.FuncAnimation(fig, draw_snapshot, frames=20, interval=200, repeat=False)
# Salva in GIF con PillowWriter
ani.save("rete_animata.gif", writer='pillow', fps=5)

Visualizzazione interattiva con Pyvis
Se vuoi esplorare uno snapshot specifico e evidenziare i link predetti, puoi usare pyvis per generare un file HTML.
Esempio: visualizza i link predetti positivi dal modello supervisionato:
from pyvis.network import Network
# === CALCOLA PROBABILITÀ ===
df_features['prob'] = clf.predict_proba(df_features[['cn', 'jc', 'aa', 'pa']])[:, 1]
# FILTRO
df_predicted = df_features[
(df_features['prob'] > 0.5) &
(~df_features.apply(lambda row: G_train.has_edge(row['u'], row['v']), axis=1))
]
# === CREAZIONE MANUALE RETE ===
net = Network(height='800px', width='100%', notebook=False, directed=False)
# Aggiungi tutti i nodi (serve esplicitamente)
for n in set(G_train.nodes()).union(df_predicted['u']).union(df_predicted['v']):
net.add_node(n, label=str(n), size=5)
# Aggiungi archi esistenti in grigio
for u, v in G_train.edges():
net.add_edge(u, v, color='gray', title='Contatto osservato', width=1)
# Aggiungi archi predetti in rosso
for _, row in df_predicted.iterrows():
net.add_edge(
row['u'], row['v'],
color='red',
title=f"Link predetto – Prob: {row['prob']:.2f}",
width=3,
dashes=True # visivamente distinguibile
)
# === VISUALIZZA ===
net.toggle_physics(True)
net.write_html("rete_predetta.html", open_browser=True)
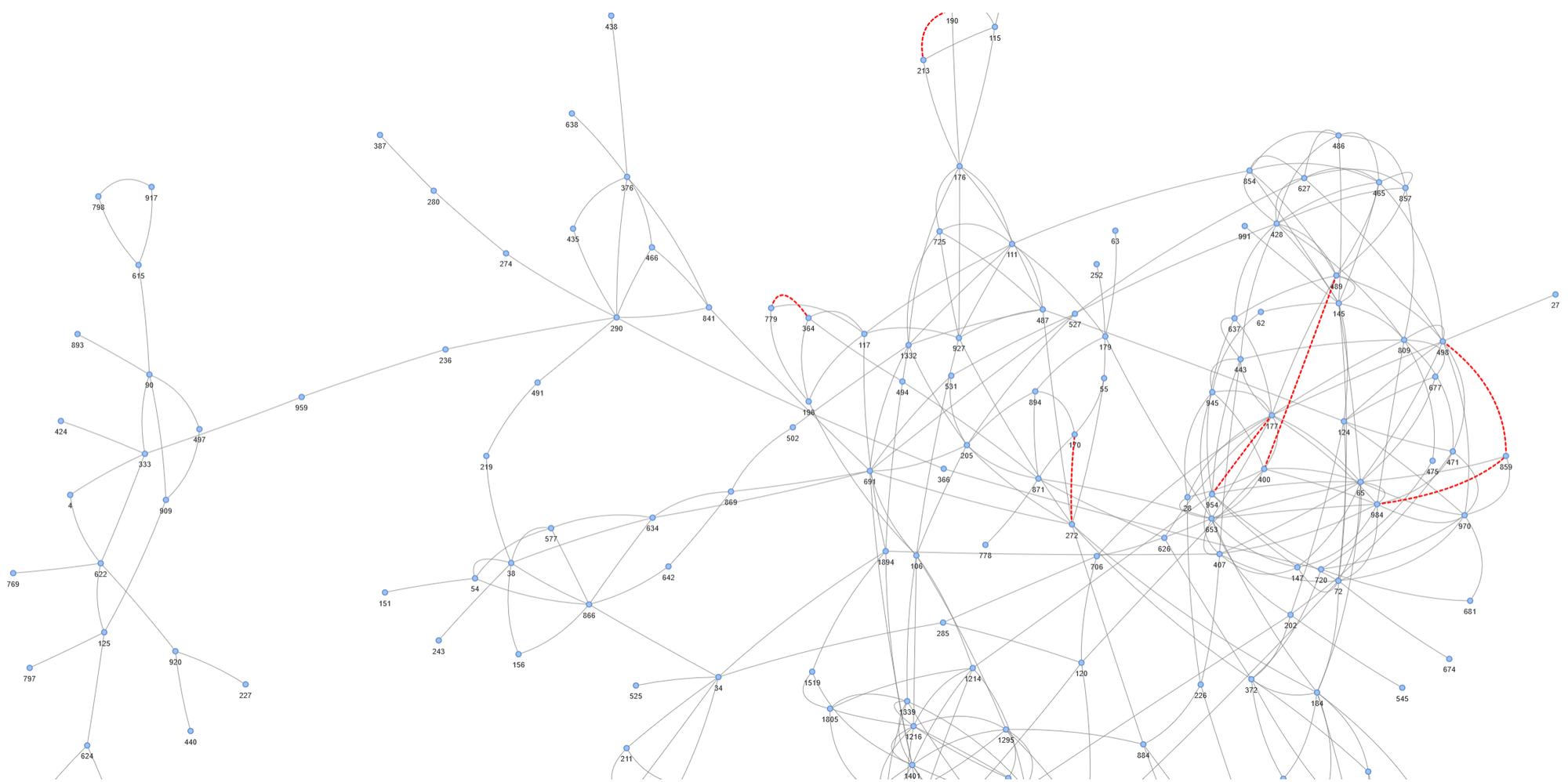
Tecniche avanzate
Quando le tecniche basate su similarità o modelli supervisionati standard non bastano (es. reti sparse, evoluzione rapida, metadati multipli), si può passare a modelli avanzati che apprendono rappresentazioni strutturali e temporali più profonde.
Embedding temporali
Gli embedding rappresentano i nodi come vettori numerici in uno spazio latente, preservando le strutture locali e globali. Esempi classici:
| Metodo | Idea principale |
|---|---|
| DeepWalk | Cammini casuali + Word2Vec |
| Node2Vec | Cammini casuali bilanciati tra BFS e DFS |
Su reti dinamiche, si applicano su ogni snapshot, producendo una sequenza di vettori che evolve nel tempo.
Esempio: Node2Vec su snapshot successivi
import networkx as nx
import pandas as pd
import numpy as np
import random
from node2vec import Node2Vec
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import roc_auc_score
# ----------------- STEP 1: Snapshot reale -----------------
print("Caricamento snapshot reale...")
G_full = snapshots[10].copy()
edges_all = list(G_full.edges())
nodes = list(G_full.nodes())
# ----------------- STEP 2: Split archi (80% train, 20% test) -----------------
print("Suddivisione archi (80% train, 20% test)...")
random.seed(42)
random.shuffle(edges_all)
split_idx = int(0.8 * len(edges_all))
edges_train = edges_all[:split_idx]
edges_hidden = edges_all[split_idx:]
G_train = nx.Graph()
G_train.add_nodes_from(nodes)
G_train.add_edges_from(edges_train)
# ----------------- STEP 3: Node2Vec su G_train -----------------
print("Addestramento Node2Vec su grafo parziale...")
node2vec = Node2Vec(G_train, dimensions=32, walk_length=10, num_walks=50, workers=2, seed=42)
model = node2vec.fit(window=5, min_count=1)
# Ciavi stringa in embedding
embedding = {str(node): model.wv[str(node)] for node in G_train.nodes()}
# ----------------- STEP 4: Coppie positive e negative -----------------
print("Generazione coppie e etichette...")
positive = [(u, v) for u, v in edges_hidden if str(u) in embedding and str(v) in embedding]
negatives = set()
while len(negatives) < len(positive):
u, v = random.sample(nodes, 2)
if not G_full.has_edge(u, v) and str(u) in embedding and str(v) in embedding:
negatives.add((u, v))
negatives = list(negatives)
pairs = positive + negatives
labels = [1] * len(positive) + [0] * len(negatives)
# ----------------- STEP 5: Feature da embedding -----------------
def edge_embedding(u, v):
return np.concatenate([embedding[str(u)], embedding[str(v)]])
X = np.array([edge_embedding(u, v) for u, v in pairs])
y = np.array(labels)
print("Totale coppie:", len(X))
print("Distribuzione etichette:")
print(pd.Series(y).value_counts())
# ----------------- STEP 6: Train/Test e RF -----------------
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, test_size=0.3, random_state=42)
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
y_pred = clf.predict_proba(X_test)[:, 1]
# ----------------- STEP 7: Risultato -----------------
auc = roc_auc_score(y_test, y_pred)
print(f"AUC finale (link prediction intra-snapshot): {auc:.4f}")
>>>
Generazione coppie e etichette...
Totale coppie: 54
Distribuzione etichette:
1 27
0 27
Name: count, dtype: int64
AUC finale (link prediction intra-snapshot): 0.6389
Reti multiplex e multiscala
Le reti reali spesso contengono più tipi di relazioni (es. amicizia, collaborazione, comunicazione) → si parla di reti multiplex.
In questo dataset, potremmo costruire:
- Layer 1: contatti fisici < 1.5 m
- Layer 2: stesse classi scolastiche (Ci == Cj)
- Layer 3 (ipotetico): relazioni dichiarate (amicizia)
Questi layer possono essere modellati separatamente o integrati con tecniche:
- Aggregazione pesata tra layer
- Embedding multilivello (uno per layer)
- GNN multiplex (es. MNE, GraphSAGE)
Esempio: grafo basato su co-classe
G_class = nx.Graph()
same_class = df[df['Ci'] == df['Cj']]
G_class.add_edges_from(zip(same_class['i'], same_class['j']))
Sfide e limiti
L’analisi di reti dinamiche è più complessa rispetto a quella delle reti statiche, sia dal punto di vista computazionale che metodologico. Di seguito le principali criticità da considerare.
1. Dati mancanti e parziali
Conseguenze:
- Bias nella struttura della rete
- Underfitting delle connessioni reali
- Link prediction che sovrastima le probabilità di contatti ripetuti
Possibili soluzioni:
- Modellare l’incertezza con approcci probabilistici
- Validare i link predetti anche in snapshot non consecutivi
- Trattare i dati mancanti come MNAR (Missing Not At Random), come discusso qui.
2. Granularità temporale
Conseguenze:
- Sovrapposizione di eventi non realmente simultanei
- Perdita della sequenza causale tra link
Possibili strategie:
- Valutare l’entropia di ogni snapshot per selezionare la dimensione ottimale
- Usare approcci multi-resolution
- Evitare discretizzazione creando modelli event-based
3. Overfitting strutturale
Conseguenze:
- Alta precisione sui dati noti
- Bassa capacità predittiva su nodi nuovi o classi rare
Soluzioni:
- Bilanciare il dataset (link reali vs non-link)
- Cross-validation su snapshot distanti
- Usare feature generalizzabili (embedding, metadati)
4. Complessità computazionale
Conseguenze:
- Tempi di calcolo lunghi
- Memoria elevata per feature e predizioni
Soluzioni:
- Campionamento negativo: selezionare un sottoinsieme casuale o “difficile”
- Parallelizzazione dei calcoli (es. embedding, scorers)
- Limitare l’analisi a componenti connesse principali
5. Interpretabilità dei modelli
Conseguenze:
- Difficoltà nel motivare le decisioni del modello
- Bassa trasparenza in contesti sensibili (es. educazione, salute, finanza)
Soluzioni:
- Feature-based modeling con interpretazione delle variabili
- Visualizzazione dei cammini predittivi
- SHAP, LIME o simili per spiegare le decisioni dei classificatori
Conclusioni
La previsione di legami in reti dinamiche è un problema centrale in molte aree applicative: epidemiologia, analisi sociale, raccomandazione di contenuti, sicurezza informatica. Questo articolo ha mostrato un approccio completo e operativo alla Link Prediction, utilizzando dati reali ad alta frequenza (High School Contact Network) e combinando tecniche classiche e avanzate di analisi delle reti.
Cosa abbiamo fatto
Abbiamo iniziato caricando un dataset di contatti a 20 secondi tra studenti, costruendo da esso una serie di snapshot temporali (reti statiche a intervalli fissi). Ogni snapshot è stato trasformato in una rete su cui abbiamo calcolato feature topologiche locali, come:
- numero di vicini comuni (Common Neighbors)
- coefficienti di similarità (Jaccard, Adamic-Adar)
- Preferential Attachment
Queste feature sono servite sia per la Link Prediction non supervisionata, sia come input per un modello supervisionato (Random Forest), addestrato a prevedere se una coppia di nodi formerà un arco nel prossimo snapshot.
Abbiamo valutato le performance con metriche appropriate per problemi sbilanciati (AUC, precision) e visualizzato i risultati con grafici statici (matplotlib) e reti interattive (pyvis).
Infine, abbiamo introdotto tecniche avanzate come gli embedding su snapshot successivi (es. Node2Vec) e discusso estensioni a reti multiplex e modelli neurali dinamici, evidenziando vantaggi, limiti e requisiti computazionali.
Principali criticità
L’analisi delle reti dinamiche presenta vincoli metodologici che vanno compresi e gestiti:
- Scelta della finestra temporale: troppo piccola → rete frammentata; troppo grande → perdita della dinamica
- Overfitting strutturale: i modelli rischiano di ripetere solo connessioni passate (effetto hub)
- Complessità computazionale: il numero di possibili link cresce quadraticamente con i nodi
- Dati mancanti: spesso i dataset catturano solo una porzione delle interazioni reali, portando a bias predittivo
Queste criticità vanno affrontate con tecniche mirate: validazione temporale, regolarizzazione o campionamento negativo. In contesti più complessi, l’integrazione con modelli neurali dinamici (es. GNN temporali) consente di modellare l’intera sequenza temporale come processo appreso, a patto di disporre di risorse computazionali adeguate.





Commenti dalla community